PARAFAC – drEEM数据前处理
这个排版,真过分,请看持续更新网址!!!
持续更新网址
语雀:语雀drEEM0.6版本
知乎专栏:有机物一些分析
数据前处理可能是耗时最长的是个一个过程,上一个专题我写到数据的前处理大概占我们平时数据处理的80%以上的时间,这个一点都不假,一定腰有耐心,慢慢来别着急,处理一次就处理好,这是后面数据分析的基础。
还是使用上次的文章的数据,这次用rawdata来做。
数据:A simple method to isolate fluorescence spectra from small dissolved organic matter datasets文中的\a_rawdata的数据
软件:MATLAB R2018a
工具箱:drEEM0.6
一、工具箱安装
首先是工具箱的安装,工具箱安装首先定位到drEEM0.6这个文件夹下,然后双击dreeminstall,光标移至编辑栏中,最后点击运行,静静等运行完,出现“Installation complete”即为安装完成

二、数据导入
2.1数据导入
部分和之前一样不再赘述,直接导入
%欢迎加入我们的讨论 群:1146318990,PARAFAC & X
cd 'D:\8.25emm\a_rawdata\rawdata'
%这里是调整在表格中荧光区域的面积,注意调整A1BQ69这个范围是包含激发和发射波长,还不是很清楚的看一下范例数据
%如果是Excel数据,改csv为Excel的后缀名。
filetype=1;ext = 'csv';RangeIn='A1..BQ69';headers=[1 1];display_opt=0;outdat=2;
[X,Emmat,Exmat,filelist_eem,outdata]=readineems(filetype,ext,RangeIn,headers,display_opt,outdat)
Ex=Exmat(1,:);
Em=Emmat(:,1);
2.2空白扣除
空白怎么扣呢,我再写一遍吧,确实是一样的,,,这个文章没有提供空白数据,我就简单哪一个数据模拟
假设我认为我有一个空白的数据测了今天所有的样本,当然这几乎是不可能的,一天估计测不完

那我有47个样本数据,那要怎么办呢,感谢cindy同学提供的建议,我在FL Solution这个软件上也看到了类似的操作,应该没有问题。我们复制47个ck文件,分别对应着剪掉不就可以了,确实可以。于是我们复制47个ck文件,ctrl+c,ctrl+v,快的不像话。

然后我们再导入这些数据,注意重命名和文件夹位置
%欢迎加入我们的讨论 群:1146318990,PARAFAC & X
cd 'D:\8.25emm\a_rawdata\ck'
filetype=1;ext = 'csv';RangeIn='A1..BQ69';headers=[1 1];display_opt=0;outdat=2;
[X_ck,Emmat,Exmat,filelist_eem_ck,outdata]=readineems(filetype,ext,RangeIn,headers,display_opt,outdat)
Ex=Exmat(1,:);
Em=Emmat(:,1);
然后我们运行这一步很简单,就是data= data荧光数据-data空白数据
X_1 = X - X_ck %因为这个空白数据是模拟的导致整个矩阵全是负值,正常情况是不会的

2.3紫外矫正
我们要在工作区中构建一个关于紫外光谱的数据集,右键新建就可以

命名为Abs并双击打开
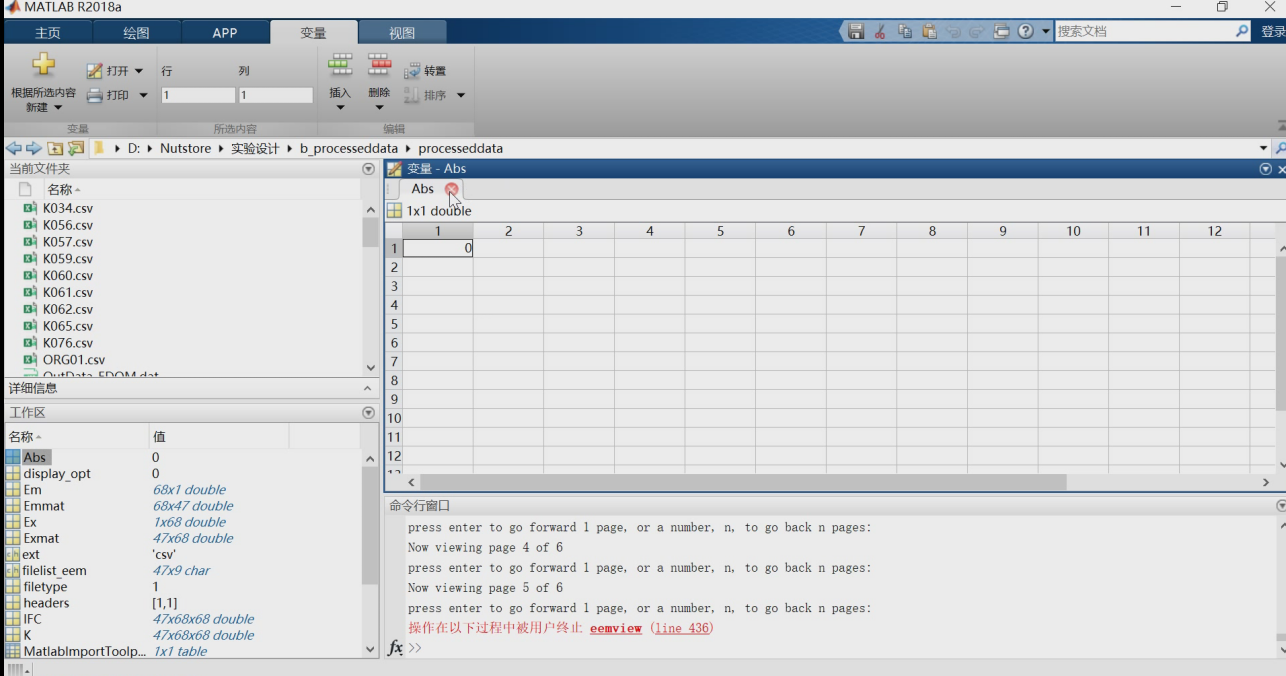
然后,在其中加入你的紫外的数据,注意,第一行为波长,第二行往下为数据,这个排列方式应该与荧光光谱的排列方式保持一致,还记得我之前很蠢的编号没,应该从001,到099这么对样本进行编号

以上工作都完成后在命令行窗口输入
[IFC,K]=ABAife(Ex,Em,Abs,X_1)
mydata= assembledataset(K,Ex,Em,'RU','longID',filelist_eem,[]);
查看绘制数据,这里我把文件名放在图上面,好知道哪个图有问题
eemview(mydata,'X',[3 3],1,[],'longID')%等会儿说参数,回车即为下一个图

以上数据整理已经进程大半,后面继续修正数据
2.4剪除拉曼瑞丽散射
我们知道,由于光线会在物质之间折射,会形成一级二级散射,这里我就不详细展开讲,我们应对这种情况的时候最常做的是是稀释物质浓度,扣除空白等,但是还是会有散射那我们应该怎么做呢?
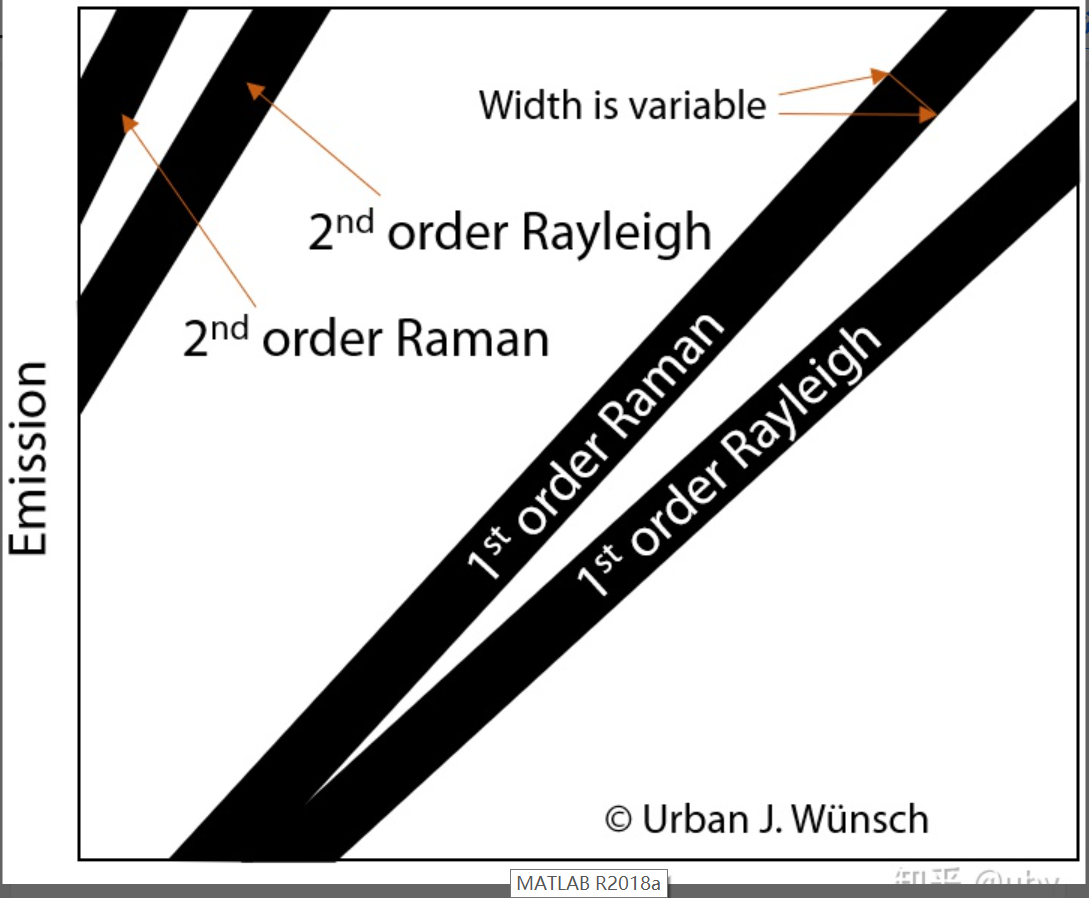
一般来讲散射区域给我们的信息并不多,一级散射可能会遮盖掉一部分的标志区(我姑且称那些发光区域为标志区)我们可以直接剪掉,如果觉得剪掉之后太丑了,我们还可以用插值拟合的办法用周围的数据填充上去,但是填充的这部分数据,指数的表达能力很糟糕。填充上去最大的目的我认为是为了后面拟合建模的时候,减少数缺失值的产生,增加模型的稳定性,当然不拟合也没关系,这篇文章中就没有拟合。
修建荧光区。这里感谢zejin同学,是zj同学从一开始就在帮我,从一些指数表达、荧光区域的选择、模型的构建等等都给我提供了很大的帮助,我第一次的实验设计、数据处理很多问题zj同学也帮助我了我很多,谢谢zj。
能给大家分享也离不开各位同学帮助,也谢谢大家。
我们一般要把荧光区域进行切割,我是说一般情况,当然整个地方我们切与不切都没关系,注意是切除后,我们把一些额外的干扰区域进行了去除
SubData=subdataset(mydata,[],mydata.Em>580,mydata.Ex<250);
eemview(SubData,'X',[3 3],1,[],'longID')
接下来我们看一下整个干扰区域到底怎么回事
eemreview(SubData)
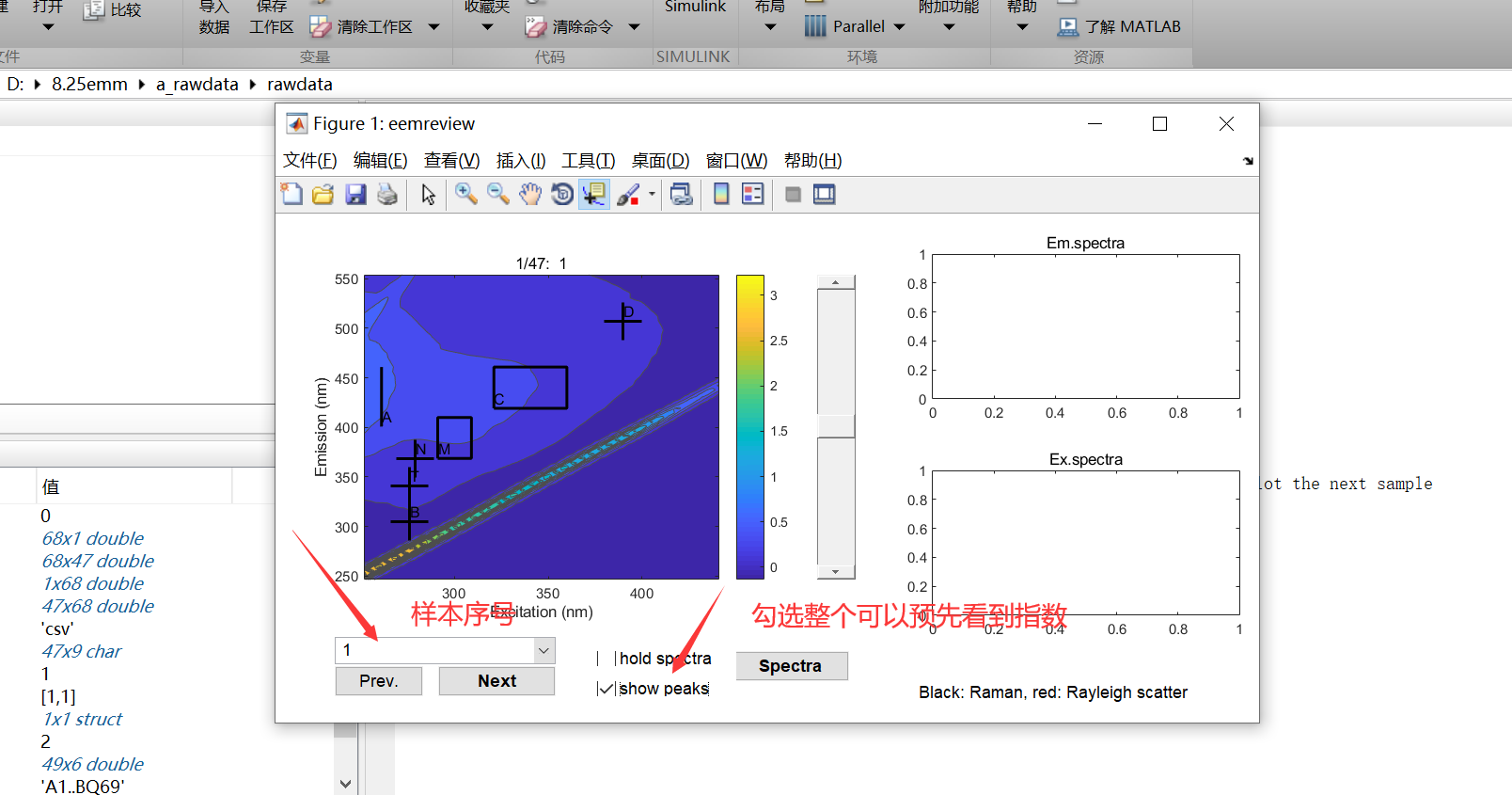
当我们点击“Spectra”的时候出现光标
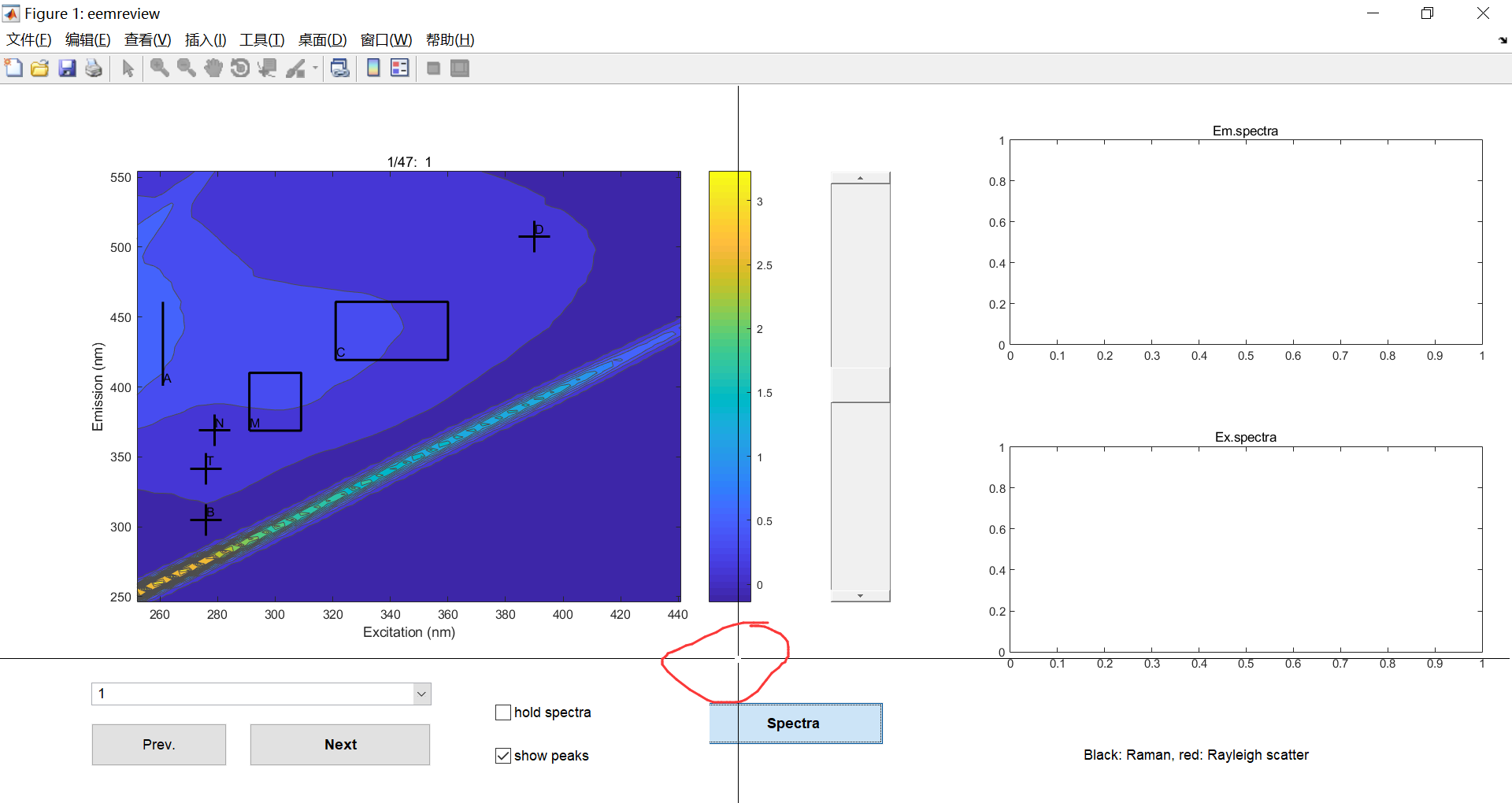
当我们把光标移动至谱图上点击之后我们看到两条线,一个灰色线一个红色线,作者的意思是,我们这个散射就是这个红色和灰色线的位置,我们要做的就是在红色和灰色线上打开缺口,换句话说,如果,散射我们剪切干净的话,粗黑线在红色和灰色线应该是断开的,等会我演示一下。
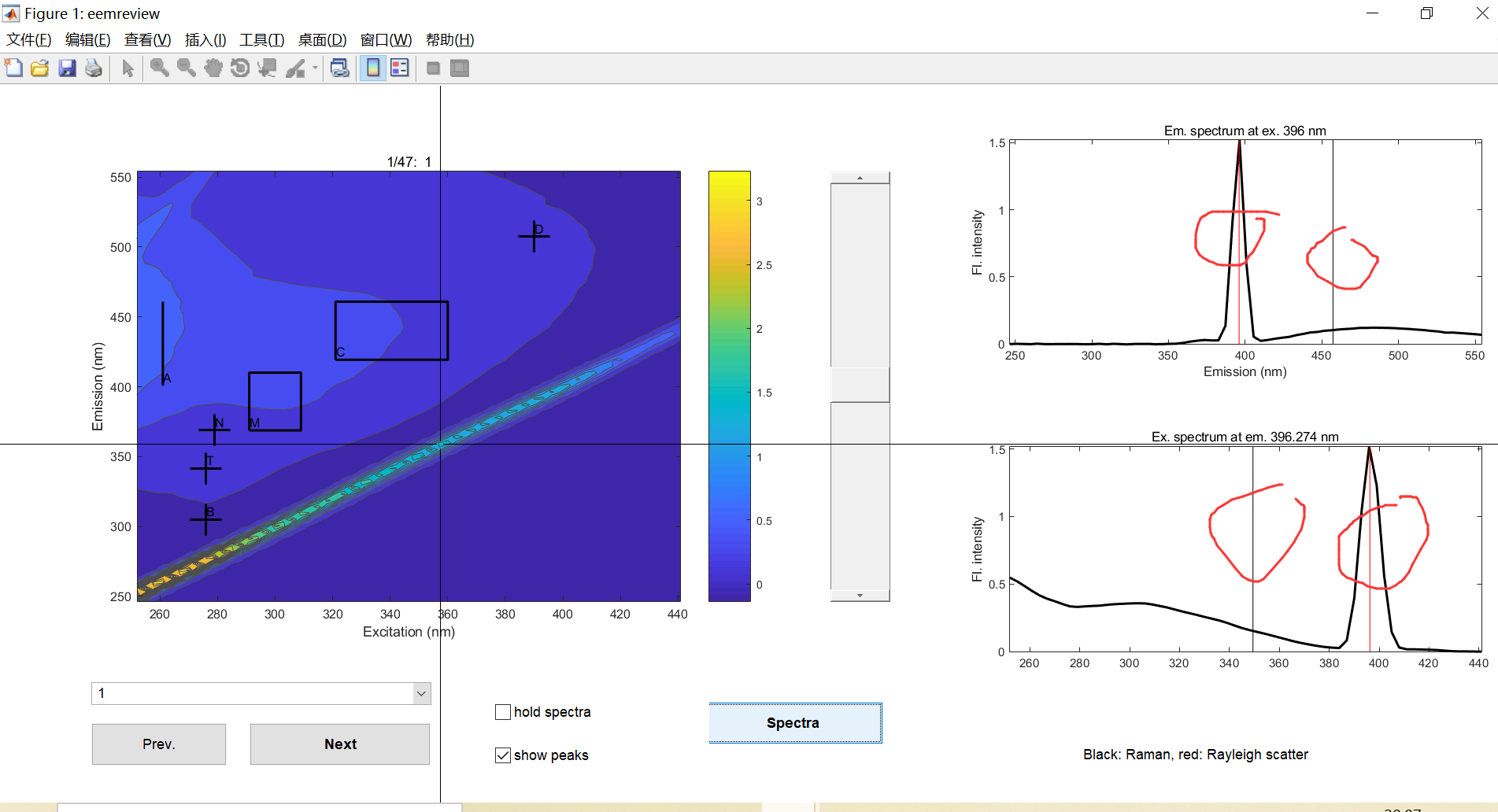
我们改一下参数,让剪切这一步在这里预先演练一下
%欢迎加入我们的讨论 群:1146318990,PARAFAC & X
eemreview(smootheem(SubData,[13 6],[5 5],[17 15],[5 5],[0 0 0 0],[],3500,0))

这里我们可以清楚的看到谁被切割了,谁有没有被切割。
我们讲一下这个参数
eemreview(smootheem(SubData,[13 20],[5 5],[17 15],[5 5],[0 0 0 0],[],3500,0))
首先SubData是我们之前的数据集,这个不多说。[13 6],[5 5],[17 15],[5 5]是怎么回事呢,[13 6],[5 5],是剪切一级瑞丽和拉曼散射,[17 15],[5 5]是剪切二级散射。数字呢,我有一个通俗的理解,你可以认为在这个散射区中间有一条线,我举1st Rayleigh为例子,在我们减去的这个散射空白中,空白上边缘距离中间的距离为13,下边缘距离为6,如此我们通过两个参数确定了散射的剪切区域。
对于我们自己的数据,不用可丁可卯的剪,只要是把散射区删除干净就可。

剪干净的标准呢,肉眼可能是最便捷的方法,当然,“spectra”按钮也看到了我们想看的结果

还有一种可能就是你这个数据很可能是其他区域也有些奇怪的点,这些点有可能就是说某个测量误差,一个小气泡之类横向也要切割应该怎么办呢

然后我输入Ex[300 320],Em[400 450]
DS=zap(SubData,1,[300 320],[400 450]);
然后再查看,嗳只是变小了,,,好吧,这确实是一个准确标点的方法,我很少用,,,
eemreview(smootheem(DS,[13 20],[5 5],[17 15],[5 5],[0 0 0 0],[],3500,0))

那么还有同学问,如果我的前一部分数据散射区域用[13 6],[5 5],[17 15],[5 5]剪切的很干净,后面一些数据就剪切不干净了怎么办,我真没什么好多办法,你把前半段数据导入后,先剪切然后导出。然后再把后半段数据导入再剪切再导出,最后合在一起。
然后还有拟合,拟合这个参数是下面这个程序的[0 0 0 0],如果改为[1 1 1 1]就把四个散射区全部拟合
eemreview(smootheem(SubData,[13 20,[5 5],[17 15],[5 5],[0 0 0 0],[],3500,0))
举例子,如果你要拟合个别散射,这四个零分别对应一级两条散射和二级两条散射,可以设为[0 1 0 1],那就是一级瑞丽和二级瑞丽不拟合,就不做演示了。
%欢迎加入我们的讨论 群:1146318990,PARAFAC & X
eemreview(smootheem(SubData,[13 20],[5 5],[17 15],[5 5],[1 1 1 1],[],3500,0))
还有最后两个参数 3500 和 0, 3500说的是能量损失频率,我不懂这个能量损失频率是什么,,,如果溶剂是水的话这个参数应该为3382. 最后一个参数0 在这里效果不明显,放在下面讲
构建剪切之后的数据集,如果最后一个参数是0,讲直接生成一个数据集,不绘制图形,如果参数改为1那就是停留1s自动绘制下一个图,如果改为’pause’注意带引号,那就是按一下空格(任意键)绘制下一个图,和DOMFluro一样,我一般’pause’,但是你数据量一旦大了,几百个肯定就不合适了,按键盘都要类似直接0算了,,
newdata=smootheem(SubData,[13 20],[5 5],[17 15],[10 10],[1 1 1 1],[],3382,'pause')
左上为原始图,右上为剪切,最下为拟合

到这里我们基本上已经把所有数据处理好了,接下来导出数据
2.5导出数据
不多说了,这里用origin,r,啥都行了
outdata1= assembledataset(newdata.X,newdata.Ex,newdata.Em,'RU','filelist',filelist_eem,[]);
Xout=outdata1;
foldername='D:\matlab\ck';%导出路径设置,这个路径一定要存在啊
for i=1:Xout.nSample
filename=deblank(char(Xout.filelist(i)));
filename=filename(1:end-3); %注意调整你末尾的位数这里-3是为了去掉csv
eem_i=squeeze(Xout.X(i,:,:));
eem_i=[[NaN; Xout.Em] [Xout.Ex'; eem_i]];
csvwrite([foldername '\' filename '_corr.csv'],eem_i)
end
matlab的绘图也很好看,细节我就不调整了
eemview(newdata,'X',[3 3],1,[],'longID',[],'wavenum','colorbar',20)%Em在纵坐标
eemview(newdata,'X',[3 3],1,[],'longID',[],'rotate','colorbar',20)%Ex在坐标轴
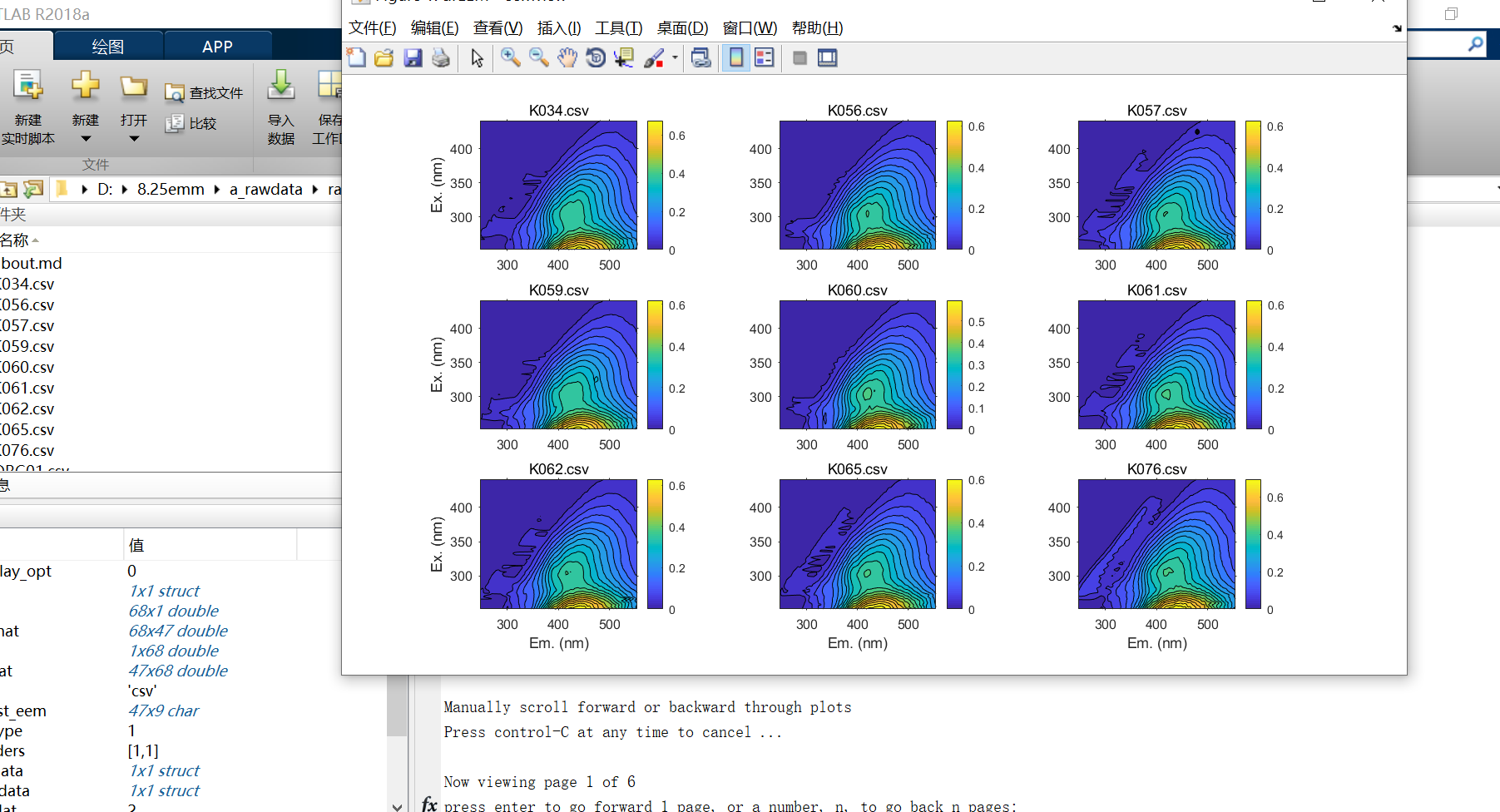
检查数据的变异性,这一步,你可大致看到所有数据的聚集情况,可以直接看到散射区有没有很干净,我不会用这个,,,如果有清楚的老师同学可以教教我啊
spectralvariance(newdata)

三、检查数据离散
使用所有数据对可能的3-6个组分数据进行拟合检验,离群值,拟合阈值很大默认是1e-2
Test1=outliertest(newdata,[],3:6);

比如你看到了9是个离散值,可以删除的话就给他删了,然后再重新运算outliertest这一步,删之前仔细画一下图看看,检查一下数据,毕竟测的都不容易,,,,
最后把newdata用在建模上
建模参考另一篇文章
————
最后,感谢我导师对我的鼓励和支持,谢谢zejin,cindy,xiaoxiao师姐,bolun,独角兽,四水,浩哥以及各位同学这一年来的帮助,谢谢各位老师的信任。
新的一年祝大家万事顺意,天天开心!
祝大家新年快乐~
以上都是基于自己的理解对PARAFAC进行归纳和总结,还有很多不对的地方,恳请各位老师同学批评指正,我的联系方式:
QQ:3414218120;邮箱:lijw092@nenu.edu.cn;微信/手机号:13844623601
2021-2-10