箱线图
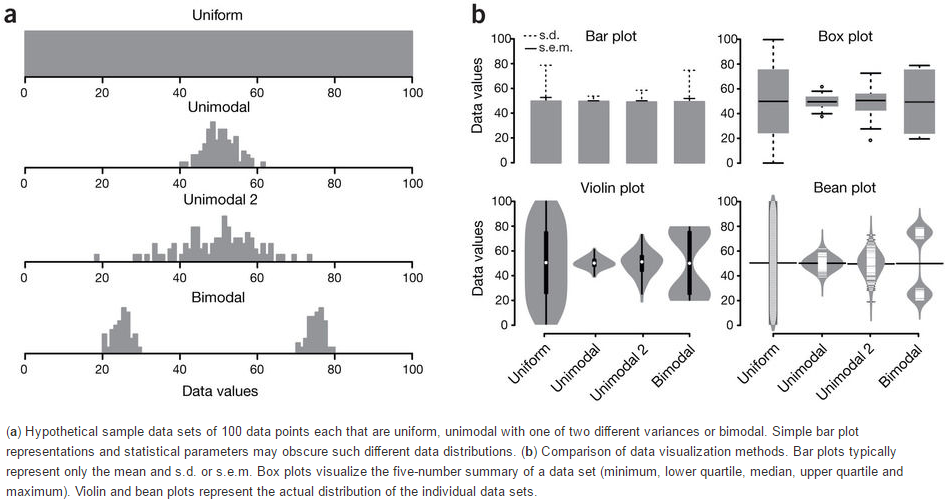
箱线图是能同时反映数据统计量和整体分布,又很漂亮的展示图。在2014年的Nature Method上有2篇Correspondence论述了使用箱线图的好处和一个在线绘制箱线图的工具。就这样都可以发两篇Nature method,没天理,但也说明了箱线图的重要意义。
下面这张图展示了Bar plot、Box
plot、Volin plot和Bean plot对数据分布的反应。从Bar plot上只能看到数据标准差或标准误不同;Box plot可以看到数据分布的集中性不同;Violin plot和Bean plot展示的是数据真正的分布,尤其是对Biomodal数据的展示。
Boxplot从下到上展示的是最小值,第一四分位数(箱子的下边线)、中位数(箱子中间的线)、第三四分位数(箱子上边线)、最大值,具体解读参见http://mp.weixin.qq.com/s/t3UTI_qAIi0cy1g6ZmHtwg。
- Nature Method文章
一步步解析箱线图绘制
假设有这么一个基因表达矩阵,第一列为基因名字,后面几列为样品名字,想绘制下样品中基因表达的整体分布。
profile="Name;2cell_1;2cell_2;2cell_3;4cell_1;4cell_2;4cell_3;zygote_1;zygote_2;zygote_3 A;4;6;7;3.2;5.2;5.6;2;4;3 B;6;8;9;5.2;7.2;7.6;4;6;5 C;8;10;11;7.2;9.2;9.6;6;8;7 D;10;12;13;9.2;11.2;11.6;8;10;9 E;12;14;15;11.2;13.2;13.6;10;12;11 F;14;16;17;13.2;15.2;15.6;12;14;13 G;15;17;18;14.2;16.2;16.6;13;15;14 H;16;18;19;15.2;17.2;17.6;14;16;15 I;17;19;20;16.2;18.2;18.6;15;17;16 J;18;20;21;17.2;19.2;19.6;16;18;17 L;19;21;22;18.2;20.2;20.6;17;19;18 M;20;22;23;19.2;21.2;21.6;18;20;19 N;21;23;24;20.2;22.2;22.6;19;21;20 O;22;24;25;21.2;23.2;23.6;20;22;21"读入数据并转换为ggplot2需要的长数据表格式(经过前面几篇的练习,这应该都很熟了)
profile_text <- read.table(text=profile,header=T, row.names=1, quote="",sep=";", check.names=F) #在melt时保留位置信息 # melt格式是ggplot2画图最喜欢的格式 #好好体会下这个格式,虽然多占用了不少空间,但是确实很方便 library(ggplot2) library(reshape2) data_m <- melt(profile_text) head(data_m)variable value
12cell_14
22cell_16
32cell_18
42cell_110
52cell_112
62cell_114
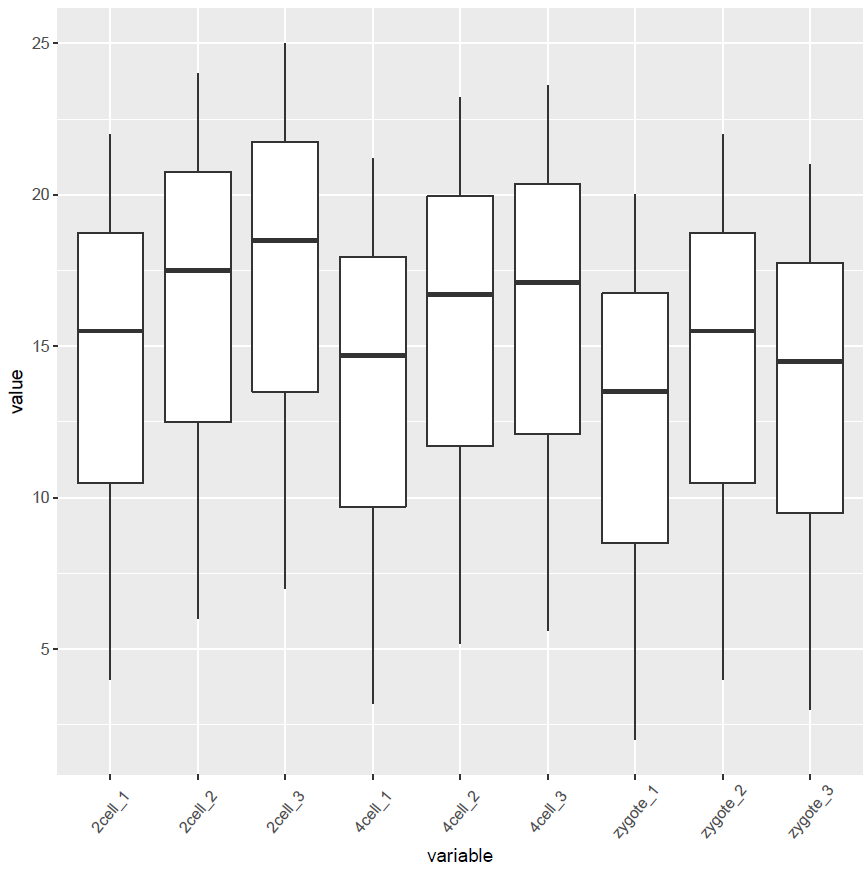
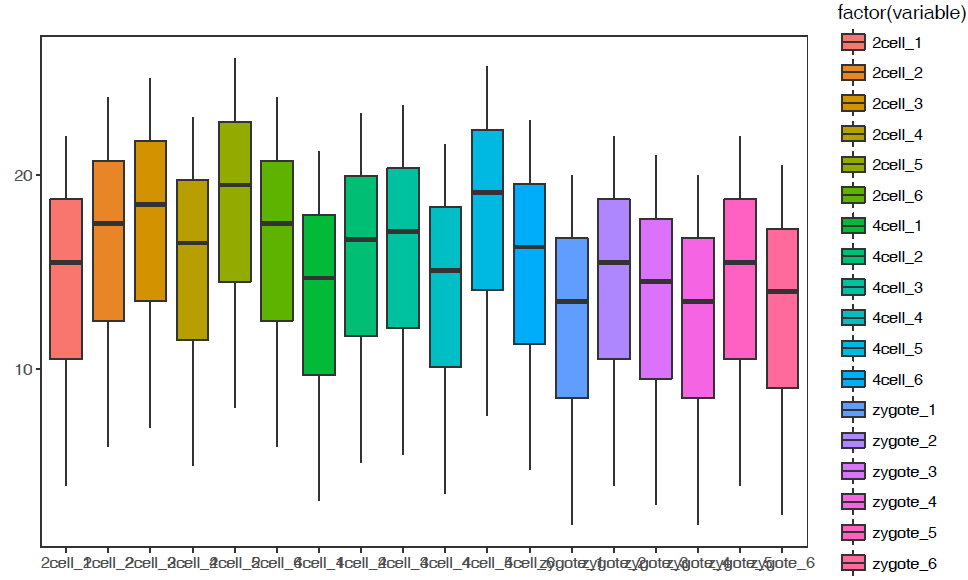
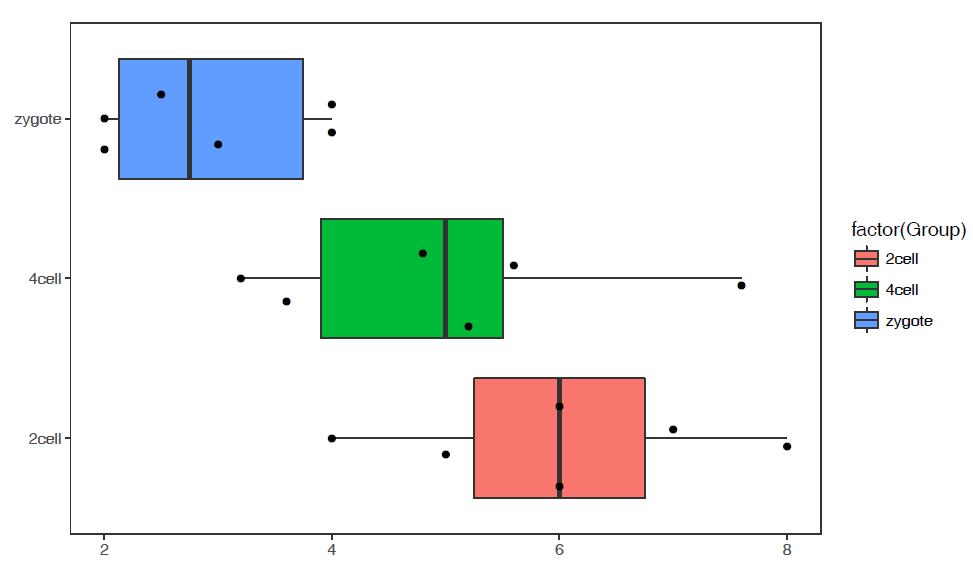
像往常一样,就可以直接画图了。# variable和value为矩阵melt后的两列的名字,内部变量, variable代表了点线的属性,value代表对应的值。 p <- ggplot(data_m, aes(x=variable,y=value),color=variable) + geom_boxplot() + theme(axis.text.x=element_text(angle=50,hjust=0.5,vjust=0.5)) + theme(legend.position="none") p #图会存储在当前目录的Rplots.pdf文件中,如果用Rstudio,可以不运行dev.off() dev.off()箱线图出来了,看上去还可以,再加点色彩。
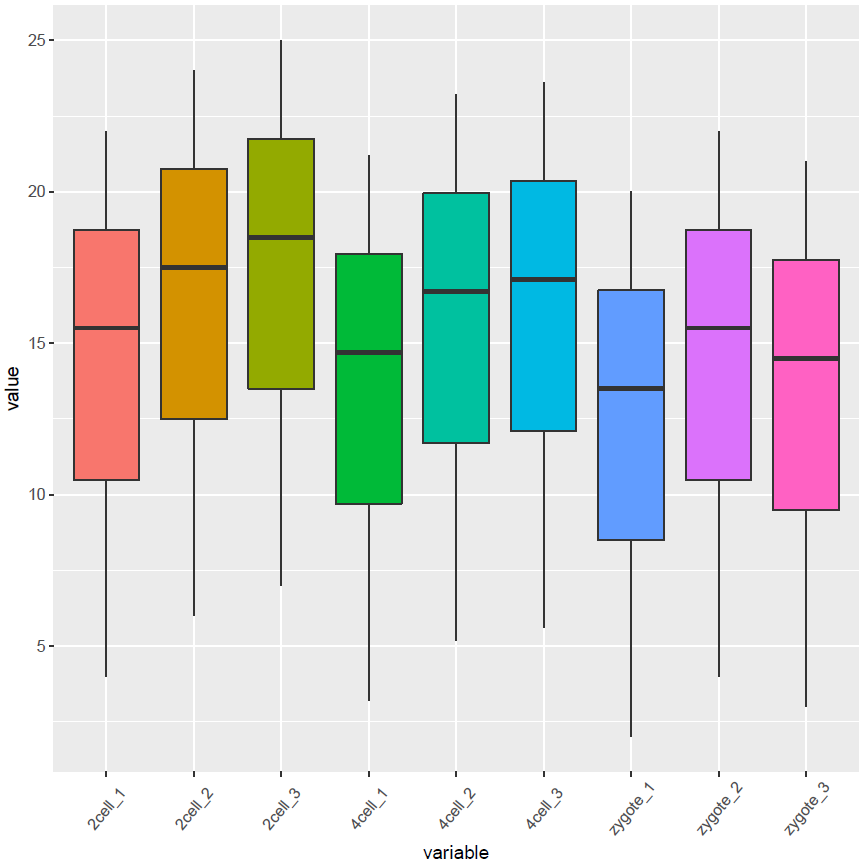
# variable和value为矩阵melt后的两列的名字,内部变量, variable代表了点线的属性,value代表对应的值。 p <- ggplot(data_m, aes(x=variable,y=value),color=variable) + geom_boxplot(aes(fill=factor(variable))) + theme(axis.text.x=element_text(angle=50,hjust=0.5,vjust=0.5)) + theme(legend.position="none") p #图会存储在当前目录的Rplots.pdf文件中,如果用Rstudio,可以不运行dev.off() dev.off()再看看Violin plot
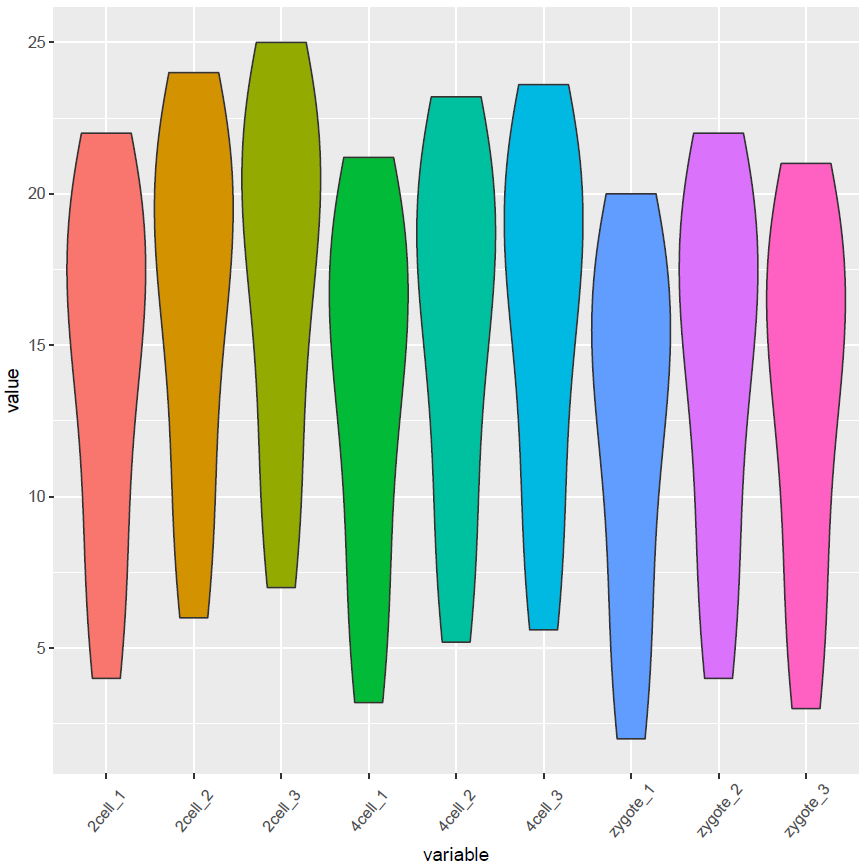
# variable和value为矩阵melt后的两列的名字,内部变量, variable代表了点线的属性,value代表对应的值。 p <- ggplot(data_m, aes(x=variable,y=value),color=variable) + geom_violin(aes(fill=factor(variable))) + theme(axis.text.x=element_text(angle=50,hjust=0.5,vjust=0.5)) + theme(legend.position="none") p #图会存储在当前目录的Rplots.pdf文件中,如果用Rstudio,可以不运行dev.off() dev.off()还有Jitter plot (这里使用的是ggbeeswarm包)
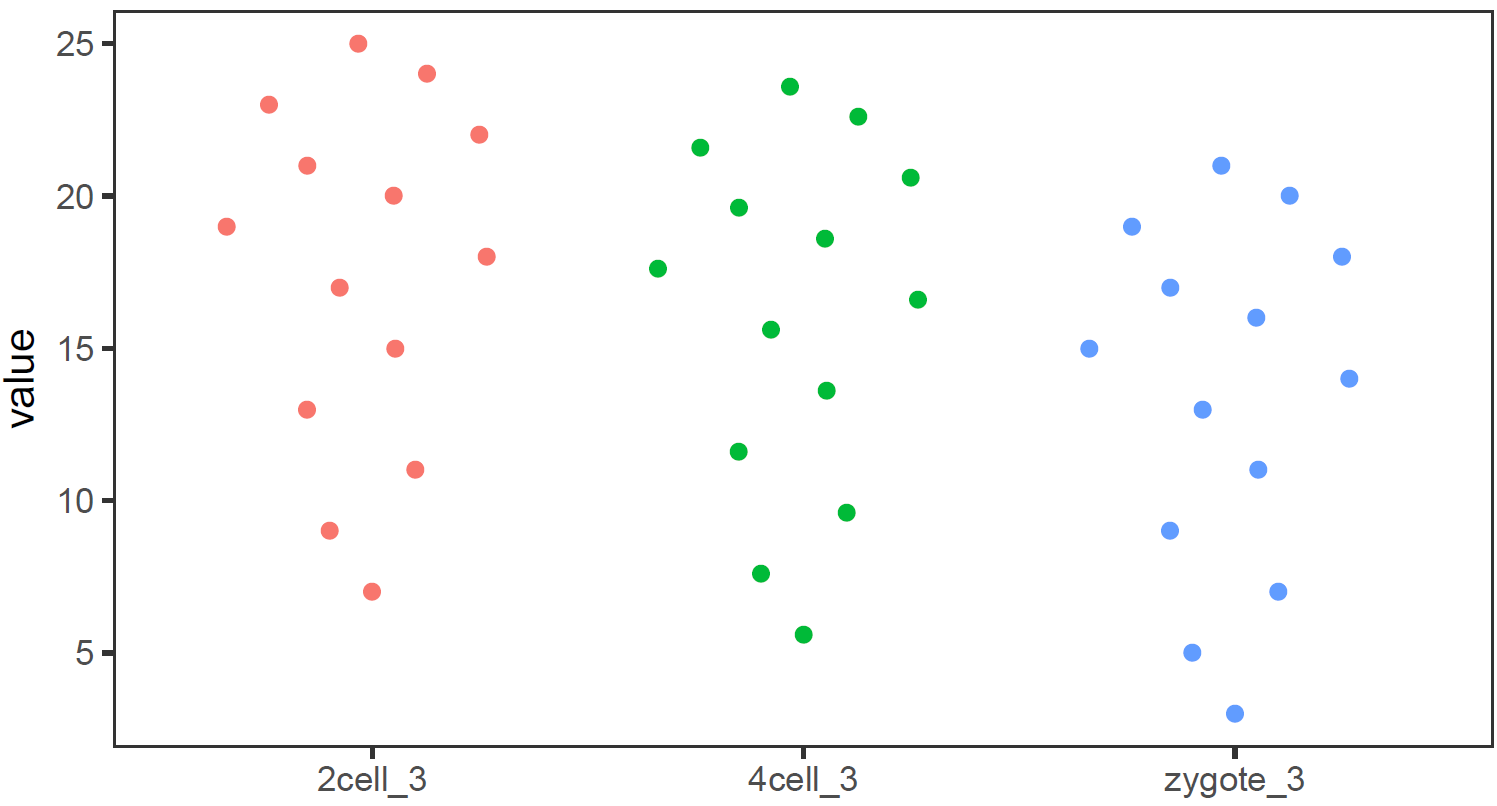
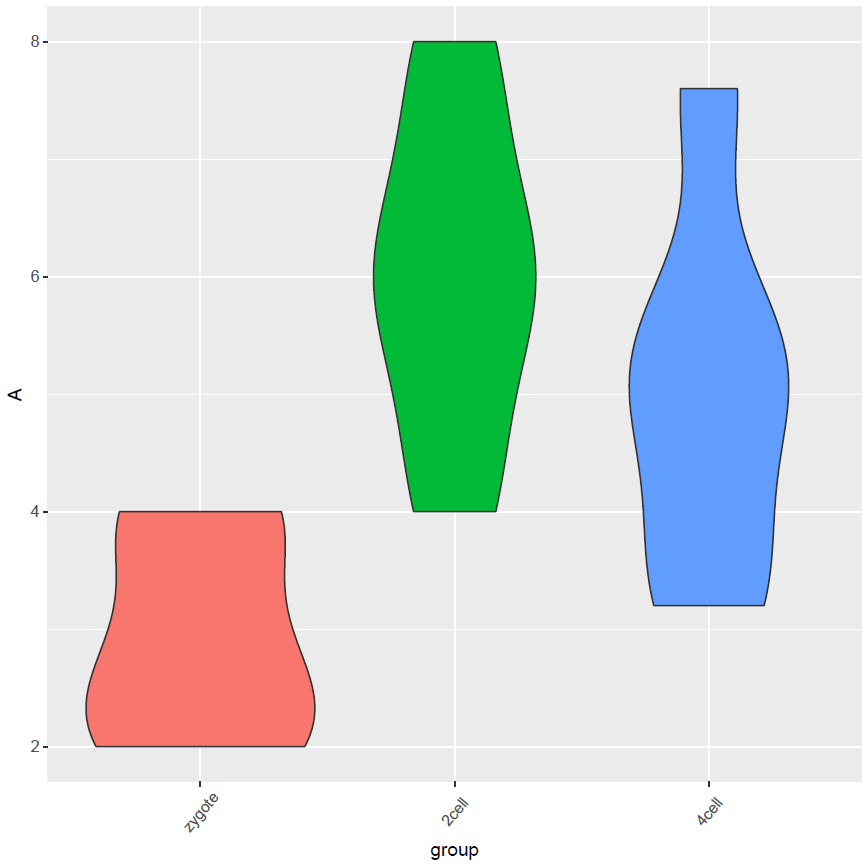
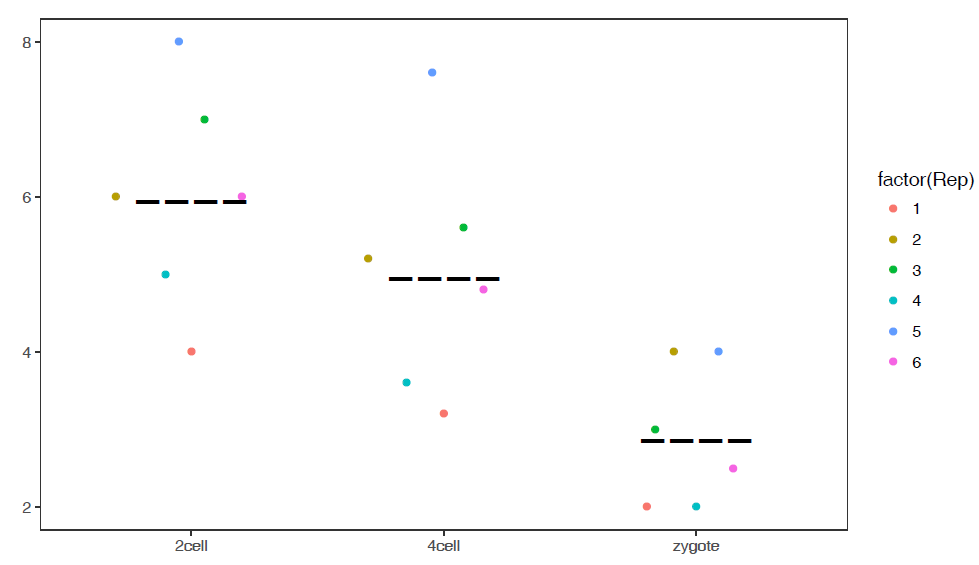
library(ggbeeswarm) #为了更好的效果,只保留其中一个样品的数据 # grepl类似于Linux的grep命令,获取特定模式的字符串 data_m2 <- data_m[grepl("_3",data_m$variable),] # variable和value为矩阵melt后的两列的名字,内部变量, variable代表了点线的属性,value代表对应的值。 p <- ggplot(data_m2, aes(x=variable,y=value),color=variable) + geom_quasirandom(aes(colour=factor(variable)))+ theme_bw() + theme(panel.grid.major =element_blank(), panel.grid.minor = element_blank(),legend.key=element_blank()) + theme(legend.position="none") #也可以用geom_jitter(aes(colour=factor(variable)))代替geom_quasirandom(aes(colour=factor(variable))) #但个人认为geom_quasirandom给出的结果更有特色 ggsave(p,filename="jitterplot.pdf", width=14, height=8,units=c("cm"))绘制单个基因(A)的箱线图
为了更好的展示效果,下面的矩阵增加了样品数量和样品的分组信息。
profile="Name;2cell_1;2cell_2;2cell_3;2cell_4;2cell_5;2cell_6;4cell_1;4cell_2;4cell_3;4cell_4;4cell_5;4cell_6;zygote_1;zygote_2;zygote_3;zygote_4;zygote_5;zygote_6 A;4;6;7;5;8;6;3.2;5.2;5.6;3.6;7.6;4.8;2;4;3;2;4;2.5 B;6;8;9;7;10;8;5.2;7.2;7.6;5.6;9.6;6.8;4;6;5;4;6;4.5" profile_text <- read.table(text=profile,header=T, row.names=1, quote="",sep=";", check.names=F) data_m = data.frame(t(profile_text['A',])) data_m$sample = rownames(data_m) #只挑选显示部分 # grepl前面已经讲过用于匹配 data_m[grepl('_[123]', data_m$sample),]Asample
2cell_14.02cell_1
2cell_26.02cell_2
2cell_37.02cell_3
4cell_13.24cell_1
4cell_25.24cell_2
4cell_35.64cell_3
zygote_12.0 zygote_1
zygote_24.0 zygote_2
zygote_33.0 zygote_3
获得样品分组信息(这个例子比较特殊,样品的分组信息就是样品名字下划线前面的部分)#可以利用strsplit分割,取出其前面的字符串 # R中复杂的输出结果多数以列表的形式体现,在之前的矩阵操作教程中 #提到过用str函数来查看复杂结果的结构,并从中获取信息 group =unlist(lapply(strsplit(data_m$sample,"_"), function(x) x[1])) data_m$group = group data_m[grepl('_[123]', data_m$sample),]Asamplegroup
2cell_14.02cell_12cell
2cell_26.02cell_22cell
2cell_37.02cell_32cell
4cell_13.24cell_14cell
4cell_25.24cell_24cell
4cell_35.64cell_34cell
zygote_12.0 zygote_1 zygote
zygote_24.0 zygote_2 zygote
zygote_33.0 zygote_3 zygote
如果没有这个规律,也可以提到类似于下面的文件,指定样品所属的组的信息。sampleGroup_text="Sample;Group zygote_1;zygote zygote_2;zygote zygote_3;zygote zygote_4;zygote zygote_5;zygote zygote_6;zygote 2cell_1;2cell 2cell_2;2cell 2cell_3;2cell 2cell_4;2cell 2cell_5;2cell 2cell_6;2cell 4cell_1;4cell 4cell_2;4cell 4cell_3;4cell 4cell_4;4cell 4cell_5;4cell 4cell_6;4cell" #sampleGroup =read.table(text=sampleGroup_text,sep="\t",header=1,check.names=F,row.names=1) #data_m <- merge(data_m, sampleGroup,by="row.names") #会获得相同的结果,脚本注释掉了以免重复执行引起问题。矩阵准备好了,开始画图了(小提琴图做例子,其它类似)
#调整下样品出现的顺序 data_m$group <- factor(data_m$group,levels=c("zygote","2cell","4cell")) # group和A为矩阵中两列的名字,group代表了值的属性,A代表基因A对应的表达值。 #注意看修改了的地方 p <- ggplot(data_m, aes(x=group,y=A),color=group) + geom_violin(aes(fill=factor(group))) + theme(axis.text.x=element_text(angle=50,hjust=0.5,vjust=0.5)) + theme(legend.position="none") p #图会存储在当前目录的Rplots.pdf文件中,如果用Rstudio,可以不运行dev.off() dev.off()长矩阵绘制箱线图
常规矩阵绘制箱线图要求必须是个方正的矩阵输入,而有时想比较的几个组里面检测的值数目不同。比如有三个组,GrpA组检测了6个病人,GrpB组检测了10个病人,GrpC组是12个正常人的检测数据。这时就很难形成一个行位检测值,列为样品的矩阵,长表格模式就适合与这种情况。
long_table <- "Grp;Value GrpA;10 GrpA;11 GrpA;12 GrpB;5 GrpB;4 GrpB;3 GrpB;2 GrpC;2 GrpC;3" long_table <-read.table(text=long_table,sep="\t",header=1,check.names=F) p <- ggplot(data_m, aes(x=Grp,y=Value),color=Grp) + geom_violin(aes(fill=factor(Grp))) + theme(axis.text.x=element_text(angle=50,hjust=0.5,vjust=0.5)) + theme(legend.position="none") p长表格形式自身就是常规矩阵melt后的格式,这种用来绘制箱线图就很简单了,就不举例子了。
箱线图-一步绘制
绘图时通常会碰到两个头疼的问题:
1.有时需要绘制很多的图,唯一的不同就是输入文件,其它都不需要修改。如果用R脚本,需要反复替换文件名,繁琐又容易出错。(R也有命令行参数,不熟,有经验的可以尝试下)
2.每次绘图都需要不断的调整参数,时间久了不用,就忘记参数怎么设置了;或者调整次数过多,有了很多版本,最后不知道用哪个了。
为了简化绘图、维持脚本的一致,我用bash对绘图命令做了一个封装,通过配置修改命令行参数,生成相应的绘图脚本,然后再绘制。
首先把测试数据存储到文件中方便调用。数据矩阵存储在boxplot.normal.data、sampleGroup和boxplot.melt.data文件中(TAB键分割,内容在文档最后。如果你手上有自己的数据,也可以拿来用)。
使用正常矩阵默认参数绘制箱线图# -f:指定输入的矩阵文件,第一列为行名字,第一行为header 列数不限,列名字不限;行数不限,行名字默认为文本 sp_boxplot.sh -f boxplot.normal.data箱线图出来了,但有点小乱。
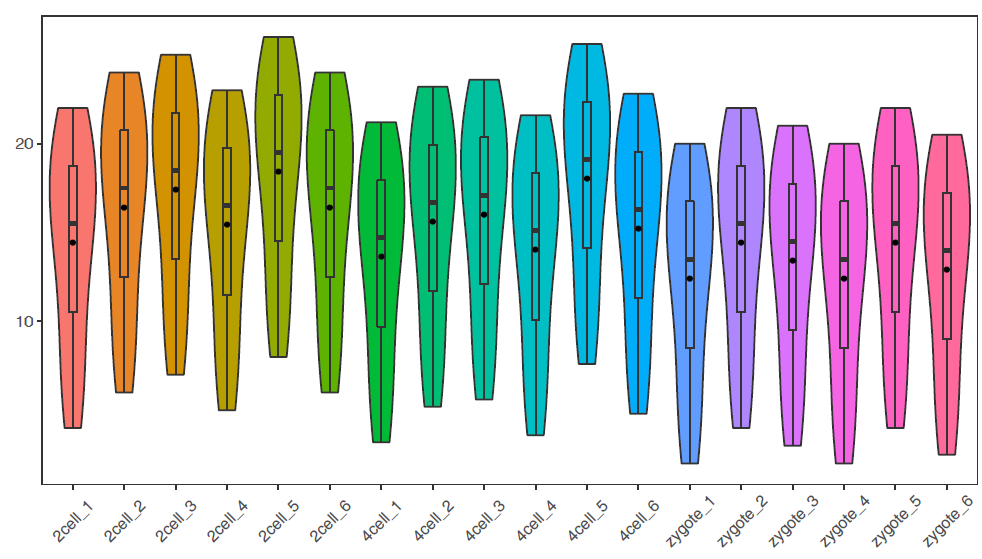
# -f:指定输入的矩阵文件,第一列为行名字,第一行为header 列数不限,列名字不限;行数不限,行名字默认为文本 # -P: none,去掉legend(uppercase P) # -b: X-axis旋转45度 # -V: TRUE绘制小提琴图 sp_boxplot.sh -f boxplot.normal.data -Pnone -b 45 -V TRUE绘制单个基因的小提琴图加抖动图
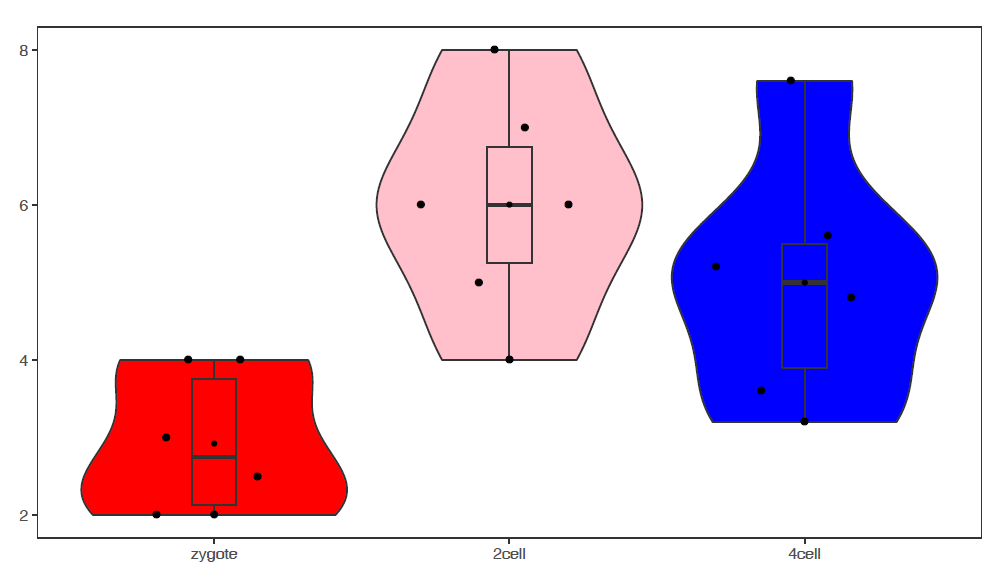
# -q:指定某一行的名字,此处为基因名,绘制基因A的表达图谱 # -Q:指定样本分组,绘制基因A在不同样品组的表达趋势 # -F Group: sampleGroup中第二列的名字,指代分组信息,根据需要修改 # -J TRUE:绘制抖动图jitter plot # -L:设置X轴样品组顺序 # -c TRUE -C "'red', 'pink', 'blue'":指定每个箱线图的颜色 sp_boxplot.sh -f boxplot.normal.data -q A-Q sampleGroup -F Group -V TRUE -J TRUE -L "'zygote','2cell','4cell'"-c TRUE -C "'red', 'pink', 'blue'" -P none使用melted矩阵默认参数绘箱线图
# -f:指定输入文件 # -m TRUE:指定输入的矩阵为meltedformat # -d Expr:指定表达值所在的列 # -F Rep:指定子类所在列,也就是legend # -a Group:指定X轴分组信息 # -j TRUE: jitter plot sp_boxplot.sh -f boxplot.melt.data -m TRUE-d Expr -F Rep -a Group-j TRUE#如果没有子类,则-a和-F指定为同一值 # -R TRUE:旋转boxplot sp_boxplot.sh -f boxplot.melt.data -m TRUE-d Expr -a Group -F Group -J TRUE -R TRUE参数中最需要注意的是引号的使用:
外层引号与内层引号不能相同 凡参数值中包括了空格,括号,逗号等都用引号括起来作为一个整体。
为了推广,也为了激起大家的热情,如果想要sp_boxplot.sh脚本的,还需要劳烦大家动动手,转发此文章到朋友圈,并留言索取。
也希望大家能一起开发,完善功能。#boxplot.normal.data Name2cell_12cell_22cell_32cell_42cell_52cell_64cell_14cell_24cell_34cell_44cell_54cell_6zygote_1zygote_2zygote_3zygote_4zygote_5zygote_6 A4675863.25.25.63.67.64.8243242.5 B68971085.27.27.65.69.66.8465464.5 C81011912107.29.29.67.611.68.8687686.5 D1012131114129.211.211.69.613.610.881098108.5 E12141513161411.213.213.611.615.612.8101211101210.5 F14161715181613.215.215.613.617.614.8121413121412.5 G15171816191714.216.216.614.618.615.8131514131513.5 H16181917201815.217.217.615.619.616.8141615141614.5 I17192018211916.218.218.616.620.617.8151716151715.5 J18202119222017.219.219.617.621.618.8161817161816.5 L19212220232118.220.220.618.622.619.8171918171917.5 M20222321242219.221.221.619.623.620.8182019182018.5 N21232422252320.222.222.620.624.621.8192120192119.5 O22242523262421.223.223.621.625.622.8202221202220.5#boxplot.melt.data GeneSampleGroupExprRep Azygote_1zygote21 Azygote_2zygote42 Azygote_3zygote33 Azygote_4zygote24 Azygote_5zygote45 Azygote_6zygote2.56 A2cell_12cell41 A2cell_22cell62 A2cell_32cell73 A2cell_42cell54 A2cell_52cell85 A2cell_62cell66 A4cell_14cell3.21 A4cell_24cell5.22 A4cell_34cell5.63 A4cell_44cell3.64 A4cell_54cell7.65 A4cell_64cell4.86#sampleGroup SampleGroup zygote_1zygote zygote_2zygote zygote_3zygote zygote_4zygote zygote_5zygote zygote_6zygote 2cell_12cell 2cell_22cell 2cell_32cell 2cell_42cell 2cell_52cell 2cell_62cell 4cell_14cell 4cell_24cell 4cell_34cell 4cell_44cell 4cell_54cell 4cell_64cellReference
*http://blog.genesino.com//2017/06/R-Rstudio
关注生信宝典,几千人一起学生信